Het wordt tijd om een alternatieve benaderingen toe te passen om de afstamming van Roechout en Boechout te onderbouwen: Genetische Genealogie. Toevallig recentelijk via mijn werk een presentatie van het Genographic project gevolgd en de laatste jaren zijn er redelijk wat vorderingen gemaakt. Ik heb twee "Paternal Lineage" testen aangevraagd (Ancestry en Family Tree DNA). Waarom twee? Ik had net de test bij Ancestry besteld en ontdekte toen Family Tree DNA ..... Deze onderneming komt wat professioneler over, dus daar ook een kit besteld. Of het verschil er daadwerkelijk is moet nog gaan blijken, want ik maak er een soort vergelijkend warenonderzoek van, dan hebben anderen daar wellicht ook wat aan.
De testen zijn nog niet binnen (nu wel, zie boven de "Family Tree DNA wang swap" van 8-1-2011), dus eerst maar wat theorie en dat ligt me goed omdat ik een moleculair-genetische achtergrond heb.
(A) Mannelijke voorouders: Y-chromosomaal DNA.
Chromosomen kaart
Ieder mens heeft in iedere lichaamscel een set aan genetische informatie (DNA) die opgeslagen is in 46 chromosomen (2 * 23). Van 22 chromosomen zijn er ieder twee paar en verder hebben vrouwen 1 paar X-chromosomen en mannen 1 X-chromosoom en 1 Y-chromosoom. Het plaatje hierboven is dus van een man. De X- en Y-chromosomen bepalen het geslacht van de nakomelingen zoals hieronder weergeven:
Er liggen een aantal genen op het Y-chromosoom die coderen voor bepaalde eigenschappen (zie figuur hierna). Deze genen blijven in de regel van generatie op generatie hetzelfde en worden niet gebruikt om genetische relaties vast te stellen.
Echter, tussen de genen liggen ook stukken DNA die niet coderen voor bepaalde eigenschappen en veranderingen in deze stukken DNA zijn wel toegestaan. Zie hieronder een overzicht van dergelijke DNA regionen op het Y-chromosoom. Het betreft (i) "Short Tandem Repeats (STR)" en (ii) "Single Nucleotide Polymorfism (SNP)".
(I) Short Tandem Repeats (STR) en directe mannelijke verwantschappen (haplotypes)
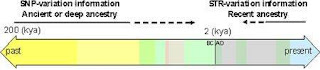
STRs kunnen worden gebruikt om relatief recente verwantschappen (tot circa 2000 jaar) tussen mannelijke lijnen aan te tonen. Alhier de theorie (bron).
De testen zijn nog niet binnen (nu wel, zie boven de "Family Tree DNA wang swap" van 8-1-2011), dus eerst maar wat theorie en dat ligt me goed omdat ik een moleculair-genetische achtergrond heb.
(A) Mannelijke voorouders: Y-chromosomaal DNA.
Chromosomen kaart
Ieder mens heeft in iedere lichaamscel een set aan genetische informatie (DNA) die opgeslagen is in 46 chromosomen (2 * 23). Van 22 chromosomen zijn er ieder twee paar en verder hebben vrouwen 1 paar X-chromosomen en mannen 1 X-chromosoom en 1 Y-chromosoom. Het plaatje hierboven is dus van een man. De X- en Y-chromosomen bepalen het geslacht van de nakomelingen zoals hieronder weergeven:
Dus het Y-chromosoom wordt doorgegeven aan de mannelijke lijn en kan gebruikt worden voor Genetische Genealogie als men de achternaam volgt. Voor het volgen van de moederlijn wordt de samenstelling van het mitochondriaal DNA (mtDNA) bestudeerd (zie verderop in dit blogartikel).
Zoals ieder chromosoom, bestaat het y-chromosoom uit twee "allelen" die met elkaar zijn verbonden (centromeer). Op ieder allel ligt een streng van (nagenoeg) identiek DNA dat in een helix is gewikkeld.
Zoals ieder chromosoom, bestaat het y-chromosoom uit twee "allelen" die met elkaar zijn verbonden (centromeer). Op ieder allel ligt een streng van (nagenoeg) identiek DNA dat in een helix is gewikkeld.
Er liggen een aantal genen op het Y-chromosoom die coderen voor bepaalde eigenschappen (zie figuur hierna). Deze genen blijven in de regel van generatie op generatie hetzelfde en worden niet gebruikt om genetische relaties vast te stellen.
Echter, tussen de genen liggen ook stukken DNA die niet coderen voor bepaalde eigenschappen en veranderingen in deze stukken DNA zijn wel toegestaan. Zie hieronder een overzicht van dergelijke DNA regionen op het Y-chromosoom. Het betreft (i) "Short Tandem Repeats (STR)" en (ii) "Single Nucleotide Polymorfism (SNP)".
(I) Short Tandem Repeats (STR) en directe mannelijke verwantschappen (haplotypes)
STRs kunnen worden gebruikt om relatief recente verwantschappen (tot circa 2000 jaar) tussen mannelijke lijnen aan te tonen. Alhier de theorie (bron).
De afkorting "DYS" staat voor: "DNA Y-chromosome Segment" en de afkorting "YSTR" staat voor "Short Tandem Repeats op het Y-chromosoom". Oftewel er zijn een aantalen regionen op het Y chromosoom (DYS) waar STRs zijn en die kunnen varieren tussen de mannelijke opeenvolgende generaties. Wat zijn Short Tandem Repeats?
Daarvoor is nog een stukje achtergrond nodig:
De genetische DNA code is opgebouwd uit vier bouwstenen. A, T, C en G. Met deze 4 elementen kan alle genetische informatie worden gemaakt. Bij "genen" wordt een combinatie van 3 bouwstenen een "codon" genoemd. Zo is het codon ATG het signaal voor de start van een gen en TAG het signaal voor de stop van een gen. Een gen dat codeert voor bijvoorbeeld de oogkleur ziet er dus alsvolgt uit.
Structuur van een gen: ATG-codon1-codon2-codon3-codon4-codon(n) - TAG
Tussen genen liggen er dus DNA fragmenten die niet belangrijk zijn en dat kunnen bijvoorbeeld korte repeterende elementen zijn.
Short Tandom Repeats: GATA-GATA-GATA-GATA-GATA-GATA-GATA-GATA-GATA-GATA-GATA
Blijkbaar kan het aantal repeterende elementen (in dit geval als voorbeeld GATA) variereren tussen opeenvolgende mannelijke generaties. Dat komt omdat er GATA fragmenten kunnen worden toegevoegd (duplicaties) of kunnen worden verwijderd (deleties). In het hierboven aangegeven voorbeeld betreft het 11 repeterende GATA eenheden op een specifieke DYS locatie. De repeterende STR eenheid noemt men ook wel een "allel".
Indien je 2 mannelijke individuen met elkaar vergelijkt, dan kun je dus uitspraken doen over een mogelijke verwantschap. Zie de tabel hieronder als voorbeeld:
Boven in de tabel staan dus een aantal regionen op het Y-chromosoom waarin variaties optreden in het aantal repeterende eenheden (DYS 393, DYS 390, etc). Het aantal repeterende eenheden (allelen) is onderzocht voor 2 individuen. Mr. Bricker 2 en Mr. Bricker 3. Voor de meeste DYS locaties blijkt het aantal allelen hetzelfde. Zo hebben beide Brickers 13 allelen bij de DYS locatie 393, oftewel er zijn in beide gevallen 13 repeterende eenheden van specifieke DNA fragmenten (bijvoorbeeld 13*GATA). Er is maar 1 afwijking bij DYS 439: Mr. Bricker 2 heeft 13 repeterende eenheden en Mr. Bricker 3 heeft 12 repeterende eenheden. Er hoeft dus slechts 1 deletie op te treden en er is dus goede kans dat beiden nauw verwant zijn. In de regel geldt dat hoe meer DYS locaties hetzelfde zijn en hoe meer DYS locaties zijn getest, de groter de kans op directe verwantschap.
Ter illustratie ook twee naamgenoten Mr. Bricker die zeer waarschijnlijk helemaal niet / veel minder verwant zijn:
Zie ook deze pagina voor achtergrond informatie in het Engels: http://blairdna.com/dna101.html
(II) Single Nucleotide Polymorfisme (SNP), evolutionaire haplogroepen en subclades.
Een tweede manier om mannelijke verwantschappen te onderzoeken zijn de SNP oftwel puntmutaties (veranderingen) op specifieke plaatsen van het Y-chromosoom. Zo kan een DNA element A in een T veranderen of een DNA element G in een C doordat er tijdens de duplicatie van het DNA een fout door het enzym DNA polymerase wordt gemaakt en deze fout wordt niet goed hersteld (wat meestal wel gebeurd).
Blijkbaar treedt de SNP variatie veel minder vaak op dan de hierboven beschreven STR variatie. Dat wil zeggen dat SNPs worden gebruikt om de wereldbevolking in specifieke groepen in te delen met als oorsprong Oost Afrika, zo'n 200.000 jaar geleden. De STR variaties beschrijven verwantschappen tot circa 2000 jaar geleden (zie ook Genebase en de presentatie in het Engels).
Een aantal van de specifieke puntmutaties die voor het bepalen van Haplogroepen worden gebruikt staan in onderstaande tabel. Ze zijn dus door verschillende laboratoria ontdekt (bron).
Er zijn momenteel 20 haplogroepen die met een letter zijn genummerd. De indeling van de groepen is bi-allellisch, dat wil zeggen een DNA bouwsteen kan van A naar T veranderen (en andersom) en van G naar C (en andersom). Zie de figuur hieronder (bron).
Binnen de haplogroepen kan een verdere onderverdeling worden gemaakt, bijvoorbeeld met behulp van STR makers (zie hierboven). Men spreekt dan van "subclades". Een gedetailleerde haplogroepkaart vind je ook op de website van Family Tree DNA (pdf downloaden duurt even).
Met behulp van de haplogroep indeling kan het verspreidingspatroon van de mensheid over de wereld in kaart worden gebracht.
en ook de verdeling van de respectievelijke haplogroepen over bijvoorbeeld Europa (bron).
R1b (rood) is dus de meest-voorkomende haplogroup in Zuid- en West Europa. Deze grote groep kan dus worden onderverdeeld in subgroepen ("subclades").
(III) Y-Chromosoom DNA sequencing.
Sinds medio 2012 is ook mogelijk om de volledige DNA volgorde van een groot gedeelte van het Y-chromosoom te laten bepalen. Met een zgn. (next generation) DNA sequencer wordt dan de volgorde van de DNA bouwstenen bepaald bijvoorbeeld: A-T-C-G-G-A-A- enzovoort. Er kunnen dan in één keer een groot aantal STRs (I) maar vooral SNPs (II) bepaald worden. Het deel van het Y-chromosoom waarvan de DNA sequentie is geanalyseerd, wordt namelijk opgenomen in een database. Het DNA wordt vergeleken met het 'standaard Y-chromosoom', zodat jouw specifieke mutaties kunnen worden vastgesteld.
Hieronder is een schematische weergave van het Y-chromosoom weergegeven inclusief het gedeelte van het Recourt dat in 2013 is gesequenced bij de firma Full Genome Corporation (https://www.fullgenomes.com). Er is van circa 20 Mbp (20 miljoen DNA baseparen) de volgorde bepaald en vergeleken met de database van bestaande en nieuwe SNPs. De verspreiding van de specifieke Recourt mutaties is als blauwe balkjes weergegeven.
Hieronder is de lijst van nieuwe "private" SNPs op het Y-chromosoom vermeld. De notaties kunnen alsvolgt worden gelezen.
kolom 1: positive van de mutatie op het standaard (b37) y-chromosoom
kolom 2: de waarde van de base op het referentie y-chromosoom (A,T,C of G)
kolom 3: de waarde van de base op het Recourt chromosoom: de mutatie of private SNP
kolom 4: de naam van de mutatie FGC staat voor Full Genome Corporation
De ruwe data van de Y-chromosoom analyse wordt opgeslagen in een zgn. .BAM file. Dit is een zeer groot bestand (enkele GB), dat ook gebruikt kan worden bij andere analyse sites. In mei 2014 is de Recourt.BAM file geanalyseerd door www.yfull.com. De Y chromosoom analyse resulteert in een complete weergave van de gevonden (gemeenschappelijke en prive) SNPs, STRs en ook het mitochondriale DNA (B, zie hieronder).
Y-chromosomale analyse kan ook bij FT-DNA worden uitgevoerd. Daar heet deze service "BigY". De analyse is goedkoper, maar er wordt ook minder DNA gesequenced. Voordeel is wel dat de analyse wordt opgenomen in de FT-DNA database, maar daar komen dus meer alternatieven, zoals YFull.
Full Genome Corporation: analyse ca. 20 - 25 Mbp, prijs medio 2014 $999,- (€ 750)
Family Tree DNAL analyse ca. 10 Mbp, prijs medio 2014 $695,- (€ 520 regelmatig aanbiedingen).
Mijn verwachting is dat de prijzen verder zullen dalen, omdat de DNA analysetechnieken steeds goedkoper worden.
(Zie ook Cruwys news)
Y-DNA Databases
Voor genealogische onderzoek komt het natuurlijk aan op de database zodat je de gegevens kunt vergelijken met reeds ingevoerde gegevens. En ik was er al bang voor: er zijn er meer dan een en ik weet nog niet of ze allemaal compatibel zijn (hetzelfde format accepteren), gekoppeld, publiek etc zijn. Zie hier een overzicht waar ik nog niet in detail naar gekeken heb: http://freepages.genealogy.rootsweb.ancestry.com/~gkbopp/DNA/YBases.htm
(B) Vrouwelijke voorouders: mitochondriaal DNA.
De samenstelling van het mitochondriaal DNA (mtDNA) is geschikt voor het volgen van de vrouwelijke genetische lijnen. Dit mtDNA wordt namelijk specifiek door de moeder overgedragen.
Bij de bevruchting versmelten de mannelijke zaadcel en de vrouwelijke eicel (bron). Zie de figuur hierboven (als je erop klikt wordt deze vergroot) en de toelichting hierna.
De mannelijke zaadcel bestaat uit een celkern met 23 (haploide) chromosomen bestaande uit 22+X of 22+Y chromsomen. De zaadcel bepaald dus het geslacht van de nakomeling (zie ook hierboven).
De vrouwelijke eicel bestaat uit een kern met 23 chromosomen (22+X = haploide) met daarom het cytoplasma waarin zich ook mitochondrien bevinden. Mitochondrien zijn eigelijk de energiefabriekjes van de menselijke cellen ("ATP" productie). Bij de mannelijke zaadcellen bevinden deze zich aan de staart en zorgen voor de energie die nodig is voor de voortbeweging van de zaadcellen.
Nadat de eicel en de zaadcel zijn versmolten bestaat de bevruchte eicel dus uit:
(1) een celkern met 23 chromosomen van de vrouw en 23 chromosomen van de man.
(2) cytoplasma afkomstig van de vrouw waain zich ook de mitochondrien van de vrouw bevinden.
In de mitochondrien bevindt zich mitochondriaal DNA (mtDNA) dat dus wordt overgedragen van de vrouwelijke stammoeder (n) naar de vrouwelijke en mannelijke nakomelingen (n+1). En dan weer van de vrouwelijke (n+1) generatie naar de vrouwelijke en mannelijke nakomelingen (n+2). Zie ook de figuur hierna (bron).
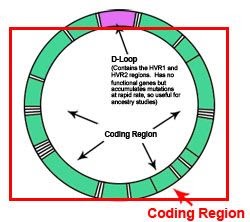
Het mt DNA is circulair en een groot gedeelte van deze cirkel bevat genen die zeer geconserveerd van generatie naar generatie worden overgedragen (zie figuur hieronder). Het betreft oa. het ATP synthase dat codeert voor een enzym betrokken bij de energieproductie (donkerblauw). Mutaties (SNPs) zijn dus zeer zeldzaam, maar komen wel voor en worden ook gebruikt voor het bepalen van mtDNA haplogroepen.
Er is ook een gedeelte dat niet codeert voor genen en mutaties komen veelvuldig voor. Het betreft de HyperVariabele regionen HV1 en HV2, bovenin de cirkel.
mtDNA analyses: HV1, HV2 en coderende regionen.
Het circulaire mitochondriaal DNA (mtDNA) bestaat dus uit 3 regionen (bron):
- Hypervariabele regio HV1
- Hypervariabele regio HV2
- Coderende regionen
Blijkbaar is een combinatie van een analyse van de niet coderende HV1, HV2 regionen en een aantal SNPs in het coderende gedeelte nodig om de oorsprong van het mtDNA te kunnen vaststellen (haplotyperingen). Vergelijkbaar bij de Y-chromosomale typering, zijn er een groot aantal maternal (26) haplogroepen gekarakteriseerd.
De basis haplogroepen worden vastgesteld dmv de SNP genotypering in de coderende regionen:
Terwijl de respectievelijke subgroepen ("subclades") kunnen worden bepaald met behulp van de DNA analyse van de HV1 (400 bp) en HV2 regionen. Een gedetailleerde kaart staat op de website van Genebase.
Net als bij Y-chromosomaal DNA kunnen de verspreidingspatronen van de respectievelijke mtDNA haplogroepen over de wereld in kaart worden gebracht. Ook hier ligt de basis in Afrika.
En tenslotte een onderverdeling voor Europa (bron):
mtDNA Databases
En ook hier een overzicht waarop verschillende mtDNA databases staan vermeld. Ik heb ze nog niet gecontroleerd:
***